特定の遺伝子をノックアウトした個体を効率よく作製する「トリプルCRISPR法」と高効率に睡眠表現型解析を行う「SSS」を開発し、新たな睡眠遺伝子「Nr3a」を発見したと、理化学研究所の上田泰己グループディレクターらの研究グループが米科学誌「Cell Reports」で発表した。
近年、特定の遺伝子を改変したりノックアウトしてどのような表現型が得られるかを観察する「逆遺伝学(リバースジェネティックス)」が注目されている。
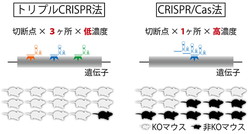
「CRISPR/Cas法」の開発によりゲノムを効率よく編集できるようになったが、一方で遺伝子のノックアウト率は約50%程度のため、ノックアウト動物を大量に作製するには数回の交配が必要である。また、複数の遺伝子を同時にノックアウトするとさらに効率が落ちるという課題もある。
研究グループは、CRISPR/Cas法を改良して、対象遺伝子の3カ所を同時に切断する「トリプルCRISPR法」を開発した。この方法をマウスに適用したところ、1世代目でほぼ100%の高い確率で大量の遺伝子ノックアウトマウスを作製することに成功した。
一方、動物の睡眠を解析するには頭蓋骨に複数の電極を装着して正確な脳波を測定する必要がある。これは手術を伴うため難易度や費用も高く、効率よく睡眠表現型の解析を行うのは困難だった。
そこで研究グループは、マウスの呼吸パターンから睡眠を解析する「SSS(Snappy Sleep Stager)」を開発した。この方法では高度な技術がいらず、コストも従来の10%以下で睡眠表現型解析が可能になった。
研究グループは、「トリプルCRISPR法」と「SSS」を用いて、さまざまな精神疾患との関連が知られる「NMDA受容体」の7つのサブユニットの遺伝子をそれぞれノックアウトしたマウスを作製、睡眠表現型解析を行った。
その結果、これまで睡眠との関係が知られていない「Nr3a」遺伝子について、ノックアウトしたマウスの睡眠時間が野生型と比べて有意に短いことがわかった。
トリプルCRISPR法は、より複雑な遺伝子改変動物を迅速に作製できる可能性があり、またSSSはヒトを含めたほかの動物についても非侵襲に測定する手法として応用が期待できるという。
(via 理化学研究所)














































 速読力をアップする方法
速読力をアップする方法
 脳力をアップする方法
脳力をアップする方法
 英語力をアップする方法
英語力をアップする方法